Mechanismus der Replikation
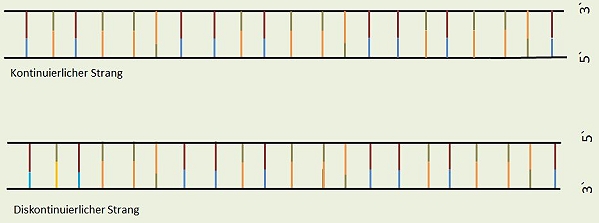
Als Okazaki Fragmente bezeichnet man kurze Abschnitte von DNA, die während der semikonservative Replikation entstehen. Meselson und Stahl haben diese Form der Verdoppelung von DNA experimentell bewiesen. Die Neubildung der DNA erfolgt an den beiden freiliegenden Strängen, die durch eine Helikase erzeugt wurden. An jedem der zwei freien Elternsträngen wird sozusagen ein Tochterstrang synthetisiert. In vielen Schemata wird dieser Vorgang auf eine bildliche Darstellung reduziert, wie sie nachstehend zu finden ist.
bbbb
bbb
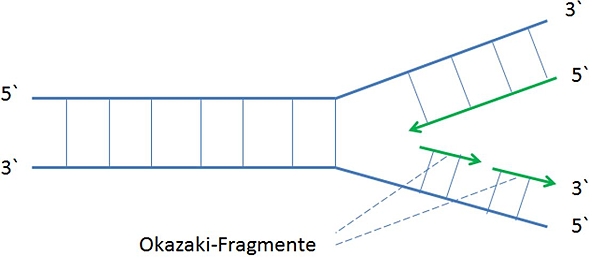
Diese ist formal korrekt, geht aber nicht auf die eigentliche Problematik der fehlenden Verknüpfung der Okazaki Fragmente untereinander durch die DNA-Polymerase ein. Betrachtet man sich die Situation genauer so erkennt man, dass die didaktische Reduktion einen wesentlichen Aspekt nicht berücksichtigt. Sowohl im kontinuierlich synthestisierten Strang als auch im diskontinuierlichen Strang erfolgt die Verknüpfung der Nukleotide oder Okazaki-Fragmente in gleicher Richtung von 3`nach 5`. Warum funktioniert dies bei den Fragmenten nicht?
bb
bb
Die hier herunterladbare Präsentation hilft Ihnen diese Abläufe, die im Folgenden erläutert werden, im Unterricht dynamisch darzustellen.
bb
bb
bb
bb
Zum Verständnis der korrekten Abläufe müssen die Schülerinnen und Schüler wissen, dass die Replikation mit Hilfe eines Multienzymkomplexes bewerkstelligt wird. Dieses besteht aus den Komponenten:
a) Helikase: Entspiralisiert die DNA
b) Primase; Stellt Primerstücke aus RNA her
c) Polymerase: Synthetisiert DNA
d) RNase: Entfernt Primer
e) Ligase Verknüpft Nukleotide
bb
bb
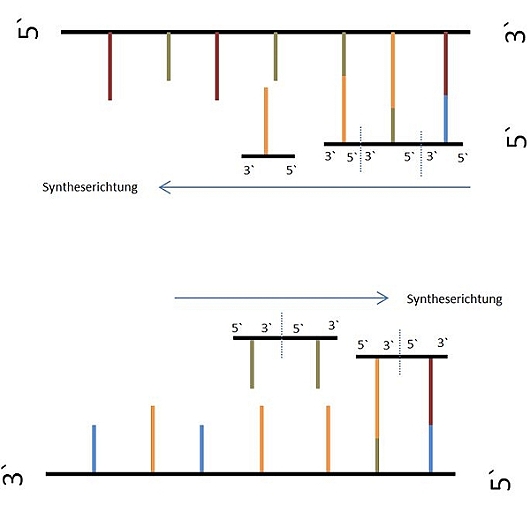
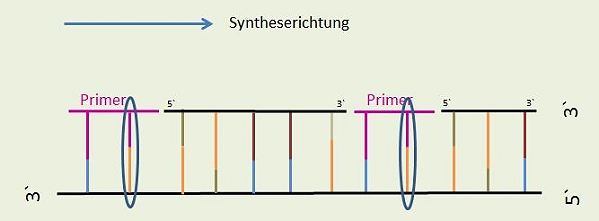
Ablauf der Replikation vom 3`Ende:
Primase synthetisiert einen Primer, der zur DNA passt, und schafft somit eine räumliche Veränderung, die durch einen Basenpaarung gekennzeichnet ist. Diese Stelle erkennt die DNA-Polymerase als Startstelle (substratspezifisch). Danach kann sie den gesamten Strang abfahren, da als Startstelle immer wieder ein Basenpaar zur Verfügung steht.
bb
bb
bb
bb
Ablauf der Replikation vom 5`Ende:
bb
Primase synthetisiert einen Primer, der zur DNA passt, und schafft somit eine räumliche Veränderung, die durch einen Basenpaarung gekennzeichtnet ist. Diese Stelle erkennt die DNA-Polymerase als Startstelle (substratspezifisch). Danach kann sie das kurze DNA-Fragment synthetisieren.
bb
bb
bb
bb
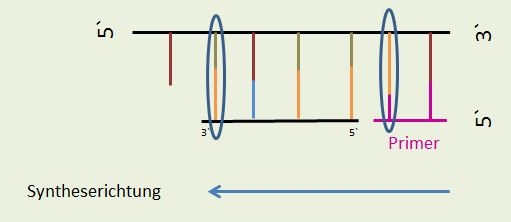
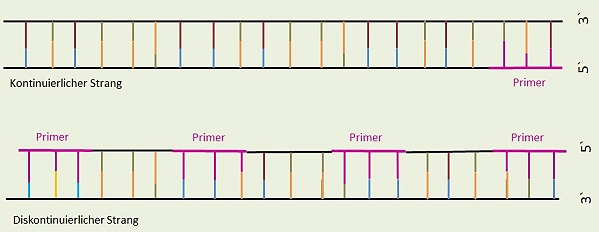
Bei jedem Neuansetzen der Polymerase muss durch die Primase erneut ein passender Primer synthetisiert werden. Die Synthese läuft dann mit Hilfe der DNA-Polymerase unter Bildung von kurzen Fragment vom neugebildeten Primer bis zum zuletzt gebildeten Primer.
Folge: Es finden sich viele Primer im diskontinuierlichen Strang.
bb
bb
bb
bb
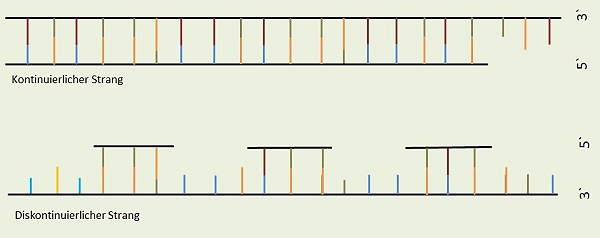
Die Primer werden durch die RNase entfernt. Nun sind auch die Okazaki- Fragmente gut sichtbar.
bb
bb
bb
Zum Schluß werden diese Lücken durch DNA-Polymerase ausgefüllt. Die Verknüpfung der Nukleotide erfolgt über eine Ligase.
bb
bb